生物学コースの竹中將起特任助教らの研究グループが環境DNAによる昆虫相の網羅的評価手法(メタバーコーディング法)を確立しました。
〜採水するだけで生息する昆虫類を網羅的に評価することが可能に〜
2024年10月18日
【研究成果のポイント】
● 産官学の連携により、種多様性の高さ故に重要でありながらも世界的課題となってきた昆虫類の環境DNA※1解析手法を確立しました。
● 河川や池の水を汲むだけで、その環境中に生息する昆虫群集を網羅的に把握可能な強力な生物モニタリング・ツールとして環境DNA解析手法を確立しました(図1)。
*ただし、本解析手法は水中だけでなく陸域の昆虫類にも適用可能です。
● 従来のミトコンドリア遺伝子(mtDNA)COI領域を対象とした昆虫類の環境DNA解析手法と比較して、本研究で新規開発した環境DNA解析手法の高い検出感度や優れた実用性が実証されました。
● 産官学の連携により、世界的にも類を見ない充実したDNAデータベースを構築しました。
● 産官学の連携により、種多様性の高さ故に重要でありながらも世界的課題となってきた昆虫類の環境DNA※1解析手法を確立しました。
● 河川や池の水を汲むだけで、その環境中に生息する昆虫群集を網羅的に把握可能な強力な生物モニタリング・ツールとして環境DNA解析手法を確立しました(図1)。
*ただし、本解析手法は水中だけでなく陸域の昆虫類にも適用可能です。
● 従来のミトコンドリア遺伝子(mtDNA)COI領域を対象とした昆虫類の環境DNA解析手法と比較して、本研究で新規開発した環境DNA解析手法の高い検出感度や優れた実用性が実証されました。
● 産官学の連携により、世界的にも類を見ない充実したDNAデータベースを構築しました。

【研究成果の具体的ポイント】
① 昆虫類のDNAバーコーディング領域として汎用されてきたCOI領域内に設計された既存の環境DNAマーカー(PCR※3プライマー※4)と、本研究において新規に提案した16S rRNA領域内に設計したマーカーを用いて、環境DNA解析における精度と検出力を比較した結果、新規マーカーの高い検出感度や優れた実用性が実証された。
従来、昆虫類のDNAバーコーディングとして使用されてきたmtDNA COI領域内に設計されたプライマーに対し、mtDNA 16S rRNA領域内に設計されたプライマーを同一の採水サンプルに対して比較解析した。この際、環境DNA解析に加えて実際の調査地における採集調査も実施した(河川水辺の国勢調査の調査法に準拠)。この結果、従来のCOIプライマーでは採集された種の半分以下の検出にとどまったのに対し、16Sプライマーでは約75%の種を検出するとともに、採集された種数の倍以上にわたる種を検出した(労力の大きな採集よりも高い効率での検出力が実証された)。
*約2割の未検出種については、現存量(バイオマス)が小さな種群である可能性が示唆され、こうした種を検出する改善策についても提唱した。
② 地球上で最も種多様性の高い昆虫類において、参照可能なDNAデータベースの充実化が遅延しているが、自治体や民間の環境コンサルタント業者との連携により、対象地域内の水生昆虫種をほぼ網羅的に登録したDNAデータベースを構築した。
精度の高い環境DNA解析を実施するために必要なDNAデータベースを構築した結果、公共のデータベースと合わせて、対象地域に生息するカゲロウ・カワゲラ・トビケラ(河川の優占種)のうち、83%(COI)、77%(16S)を登録した(特異なハビタットに生息するような種を除き、一般的な河川調査において検出されるような種群については、ほぼ網羅的に登録された)。
【研究の背景と研究手法】
水資源の保全と管理は、人々が快適で豊かな生活を維持するために重要かつ不可欠ですが、人間活動により淡水の半分以上が利用されています。その結果として、生物多様性が最も急速に減少しているのが淡水生態系であると言われています。
水質の変化や環境の健康状態に敏感である水生昆虫類は、水質の指標として利用され(Cain et al., 1992; Susmita et al., 2013)、その長期的な種多様性や遺伝的多様性のモニタリングは、河川生態系の保全だけでなく、水質の管理にも有用です(Costanza & Mageau, 1999; Sakata et al., 2022)。従来の捕獲を伴う調査には多大な労力と時間や費用を要するのに対し、近年では、多くの生態系における水生生物の生物多様性を簡単かつ効果的なモニタリングを可能とする環境DNA(eDNA)に焦点を当てた調査研究が注目を集めています(Bohmann et al., 2014; Miya et al., 2015; Deiner et al., 2017)。さらに、環境DNA解析は、生物の種多様性の評価に加えて、絶滅危惧種の遺伝的多様性を非侵襲的に評価することができるのが特長です(Ahn et al., 2020; Sekiya et al., 2017)。
環境DNAとは、環境中(河川や湖、海、土壌、空気など)に浮遊しているDNAを指します。これらの環境DNAを捕捉し、DNAバーコーディング法に基づく分子種同定を網羅的に実施することで、環境中に生息している生物群集を把握するのがメタバーコーディング法であり、近年注目を集めている画期的な手法です。
しかし昆虫類においては、環境DNA解析の効果的手法が確立されていないのが現状であり、急務の課題でした。特に、世界的に汎用されているmtDNA COI領域を用いた環境DNA解析では、網羅的な種の検出が困難でること、解析の対象ではない分類群(珪藻や藻類など)のDNAが増幅されてしまうなどの問題がありました。そうしたなか、我々は先行研究(Takenaka et al., 2023)において、環境DNA解析における技術面で先行する魚類や両生類などの研究事例を参考に、新規DNAバーコーディング領域をmtDNA 16S rRNA領域内に設計することを提案し、新規PCRプライマー「MtInsects-16S」を開発しました。このプライマーは、昆虫類において高い汎用性、つまりほぼ全ての昆虫種のDNAを増幅することのできるプライマーである可能性を示唆しました。
こうした背景下、本研究では、この新規開発した環境DNAのメタバーコーディング・マーカーを実際に野外で利用することで、その有用性を検証しました。この際、世界的によく使われているミトコンドリア遺伝子COI領域のマーカーと、我々が開発したMtInsects-16Sの両方のマーカーを用いて、環境DNA解析を実施しました。また同時に、同一地点において実際に採集調査も実施することで、3つの方法による解析結果を比較しました。
① 昆虫類のDNAバーコーディング領域として汎用されてきたCOI領域内に設計された既存の環境DNAマーカー(PCR※3プライマー※4)と、本研究において新規に提案した16S rRNA領域内に設計したマーカーを用いて、環境DNA解析における精度と検出力を比較した結果、新規マーカーの高い検出感度や優れた実用性が実証された。
従来、昆虫類のDNAバーコーディングとして使用されてきたmtDNA COI領域内に設計されたプライマーに対し、mtDNA 16S rRNA領域内に設計されたプライマーを同一の採水サンプルに対して比較解析した。この際、環境DNA解析に加えて実際の調査地における採集調査も実施した(河川水辺の国勢調査の調査法に準拠)。この結果、従来のCOIプライマーでは採集された種の半分以下の検出にとどまったのに対し、16Sプライマーでは約75%の種を検出するとともに、採集された種数の倍以上にわたる種を検出した(労力の大きな採集よりも高い効率での検出力が実証された)。
*約2割の未検出種については、現存量(バイオマス)が小さな種群である可能性が示唆され、こうした種を検出する改善策についても提唱した。
② 地球上で最も種多様性の高い昆虫類において、参照可能なDNAデータベースの充実化が遅延しているが、自治体や民間の環境コンサルタント業者との連携により、対象地域内の水生昆虫種をほぼ網羅的に登録したDNAデータベースを構築した。
精度の高い環境DNA解析を実施するために必要なDNAデータベースを構築した結果、公共のデータベースと合わせて、対象地域に生息するカゲロウ・カワゲラ・トビケラ(河川の優占種)のうち、83%(COI)、77%(16S)を登録した(特異なハビタットに生息するような種を除き、一般的な河川調査において検出されるような種群については、ほぼ網羅的に登録された)。
【研究の背景と研究手法】
水資源の保全と管理は、人々が快適で豊かな生活を維持するために重要かつ不可欠ですが、人間活動により淡水の半分以上が利用されています。その結果として、生物多様性が最も急速に減少しているのが淡水生態系であると言われています。
水質の変化や環境の健康状態に敏感である水生昆虫類は、水質の指標として利用され(Cain et al., 1992; Susmita et al., 2013)、その長期的な種多様性や遺伝的多様性のモニタリングは、河川生態系の保全だけでなく、水質の管理にも有用です(Costanza & Mageau, 1999; Sakata et al., 2022)。従来の捕獲を伴う調査には多大な労力と時間や費用を要するのに対し、近年では、多くの生態系における水生生物の生物多様性を簡単かつ効果的なモニタリングを可能とする環境DNA(eDNA)に焦点を当てた調査研究が注目を集めています(Bohmann et al., 2014; Miya et al., 2015; Deiner et al., 2017)。さらに、環境DNA解析は、生物の種多様性の評価に加えて、絶滅危惧種の遺伝的多様性を非侵襲的に評価することができるのが特長です(Ahn et al., 2020; Sekiya et al., 2017)。
環境DNAとは、環境中(河川や湖、海、土壌、空気など)に浮遊しているDNAを指します。これらの環境DNAを捕捉し、DNAバーコーディング法に基づく分子種同定を網羅的に実施することで、環境中に生息している生物群集を把握するのがメタバーコーディング法であり、近年注目を集めている画期的な手法です。
しかし昆虫類においては、環境DNA解析の効果的手法が確立されていないのが現状であり、急務の課題でした。特に、世界的に汎用されているmtDNA COI領域を用いた環境DNA解析では、網羅的な種の検出が困難でること、解析の対象ではない分類群(珪藻や藻類など)のDNAが増幅されてしまうなどの問題がありました。そうしたなか、我々は先行研究(Takenaka et al., 2023)において、環境DNA解析における技術面で先行する魚類や両生類などの研究事例を参考に、新規DNAバーコーディング領域をmtDNA 16S rRNA領域内に設計することを提案し、新規PCRプライマー「MtInsects-16S」を開発しました。このプライマーは、昆虫類において高い汎用性、つまりほぼ全ての昆虫種のDNAを増幅することのできるプライマーである可能性を示唆しました。
こうした背景下、本研究では、この新規開発した環境DNAのメタバーコーディング・マーカーを実際に野外で利用することで、その有用性を検証しました。この際、世界的によく使われているミトコンドリア遺伝子COI領域のマーカーと、我々が開発したMtInsects-16Sの両方のマーカーを用いて、環境DNA解析を実施しました。また同時に、同一地点において実際に採集調査も実施することで、3つの方法による解析結果を比較しました。
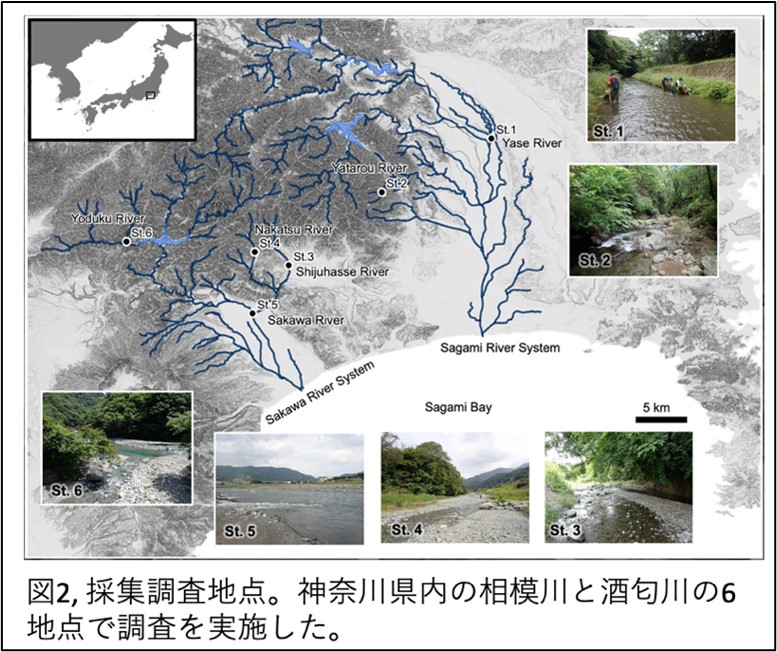
昆虫類において網羅的な分子種同定(DNAメタバーコーディング)を実施する際には、DNAデータベースの情報不足による解析バイアスの問題も懸念されていますが、調査に先駆けて、調査対象地域の水生昆虫(特にカゲロウ類・カワゲラ類・トビケラ類)に関するDNAデータベースを独自に構築しました(公共データベースへの登録も済み)。DNAデータベースの構築については、神奈川県環境科学センターが中心となり、株式会社プラントビオと株式会社生物技研の協力のもとに実施されました。
実際の調査に関しては、神奈川県内の相模川水系および酒匂川水系内に6つの調査地点を設定し、各地点で3回ずつ1Lの採水を実施し、同時に水生昆虫の採集(定量調査+定性調査)も実施しました。採水したサンプルは、mtDNA COIおよび16S rRNA領域に基づく環境DNA分析を実施しました。
これらの結果を、同じサイトでの物理的捕獲調査と比較し、確立されたデータベースを使用して昆虫目の検出能力を検証しました。
実際の調査に関しては、神奈川県内の相模川水系および酒匂川水系内に6つの調査地点を設定し、各地点で3回ずつ1Lの採水を実施し、同時に水生昆虫の採集(定量調査+定性調査)も実施しました。採水したサンプルは、mtDNA COIおよび16S rRNA領域に基づく環境DNA分析を実施しました。
これらの結果を、同じサイトでの物理的捕獲調査と比較し、確立されたデータベースを使用して昆虫目の検出能力を検証しました。
【結果・考察】
環境DNA解析に先駆けて、神奈川県の水生昆虫のDNAデータベースを構築した結果、公共のデータベースであるNCBIと組み合わせると、神奈川県内に記録されている種の中で、mtDNA 16S rRNA領域で77%、ミトコンドリアDNA COI領域で83%の種を登録することができました。特異的なハビタットに生息するような種などが未登録の状況にはあるなど完全なではないものの、世界的にもここまで充実したDNAデータベースはなく、データベースの情報不足による評価バイアスは最小限に抑えられました。
環境DNA解析に先駆けて、神奈川県の水生昆虫のDNAデータベースを構築した結果、公共のデータベースであるNCBIと組み合わせると、神奈川県内に記録されている種の中で、mtDNA 16S rRNA領域で77%、ミトコンドリアDNA COI領域で83%の種を登録することができました。特異的なハビタットに生息するような種などが未登録の状況にはあるなど完全なではないものの、世界的にもここまで充実したDNAデータベースはなく、データベースの情報不足による評価バイアスは最小限に抑えられました。
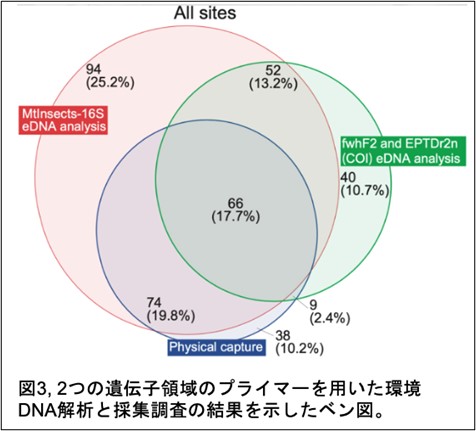
環境DNA解析の結果、我々が開発したmtDNA 16S rRNA領域内に設計されたMtInsects-16Sプライマーを用いた場合に最も多くの種が検出されました。採集調査で実際に採集した種の74.9%がMtInsects-16Sマーカーにより検出され(mtDNA COI領域のプライマーセットでは40.1%のみの検出)、採集調査(178種)では確認できなかった146種を含む286種が検出されました(採集調査の1.6倍)。また、COI領域のマーカーでのみ検出された種は、河川内でのバイオマスが小さい可能性が示唆され、MtInsects-16SマーカーによるDNA解析の精度と検出力の高さを実証しました。
本研究では、産官学連携により、調査対象とした地域特有のDNAバーコーディング※2用データベースを整備したことも特筆するべき成果と言えます。このデータベースは世界的にも類を見ない整備レベルであり、結果として、より高い精度での群集構造が評価できました。
本研究では、産官学連携により、調査対象とした地域特有のDNAバーコーディング※2用データベースを整備したことも特筆するべき成果と言えます。このデータベースは世界的にも類を見ない整備レベルであり、結果として、より高い精度での群集構造が評価できました。
【波及効果、今後の課題】
環境DNA解析は、水を汲み、濾過しただけの試料から、その環境に生息している対象とする生物相を把握することができる強力な生物モニタリング・ツールです。特定の環境における生物多様性の評価や、絶滅危惧種の生息の有無、その遺伝的多様性を評価する際にも有効です。水を汲むだけの非侵襲的サンプリングであり、生態系への影響も最小限です。
また、我々が開発したマーカーは、採水サンプルからの環境DNA解析(メタバーコーディング)による生物相の評価だけでなく、例えば、魚類の胃内容物や哺乳類の糞便試料から対象とする魚類や哺乳類の食性を網羅的に把握するようなメタバーコーディング解析にも応用可能であり、既に、こうした調査研究においても用いられ、強力なツールとして効果を発揮しています。本研究でも、陸生昆虫が多く検出されており、水生昆虫だけでなく、陸生昆虫を含めた昆虫類全般を対象とした網羅的解析も可能です。こうしたことから、近い将来におけるメタバーコーディング手法として汎用されることが期待されます。
【関連する論文】
本研究成果の筆頭著者・責任著者らによる先行研究(Takenaka M, Yano K, Suzuki T, Tojo K, 2023, Development of novel PCR primer sets for DNA barcoding of aquatic insects, and the discovery of some cryptic species, Limnology, 24: 121–136)において、昆虫類に汎用可能であるメタバーコーディング・マーカーを新規開発しており、本研究ではそのマーカーを実践的研究に用いることで、実用性の高さを実証した関係にある。Takenaka et al. (2023) は、論文公表以来、2000ダウンロードを超える多くのアクセスや引用(現時点で10論文に引用)があり、これらの反響の大きかさら、2024年10月に Limnology 誌の論文賞(Limnology Excellent Paper Award)を受賞しました。
【用語解説】
※1. 環境DNA 河川や湖沼などの環境中に浮遊する生物由来のDNAをさす。こうした環境中のDNAを解析することで、その環境中に生息している生物相を把握することができる。特定の種を対象に、対象種の生息の有無を評価する方法や、今回のように対象分類群(群集)を網羅的に解析する方法などが挙げられる。
※2. DNAバーコーディング 「標準化」された遺伝子領域(DNA断片)を対象に、データベースの登録配列を参照することで、迅速、正確、そして自動的に種を同定する概念。スーパーマーケットの製品コードである黒い縞模様(バーコード)をスキャナーで読み取る方法と似ていることから名付けられたもので、高い専門知識がなくても種同定が可能になるだけでなく、昆虫類の場合、幼虫や卵、不完全な標本(体の一部分など)からも種同定することができる。しかし、高い精度の分類学的根拠に基づくDNAデータベースの構築が必須。
※3. PCR ポリメラーゼ連鎖反応(Polymerase Chain Reaction)の略称で、対象とする遺伝子配列を増幅する手法。わずかなDNA量のサンプルであっても、特定の配列だけを様々な解析に使用できるように増幅させる。新型コロナウイルスの感染の有無を判定する際にも同じメカニズムを用いて、PCRによりウィルス特定の配列が増幅されるかどうかが検査された。
※4. PCRプライマー PCRにより特定のDNA配列を増幅させる際に、増幅させる遺伝子配列の両端に結合させて、DNAの増幅の開始点として機能させる20塩基程度の短い遺伝子断片。DNAに結合した2つのプライマーが、結合した箇所から互いにDNAの複製が進むことで、2つのプライマーの内側の配列が増幅される。新型コロナウイルスの感染の有無を検査するPCRにおいては、このウイルスに特異的なプライマーを設計することで、体内にウイルスが存在している時にのみその特定遺伝子配列が増幅される仕組みが利用された。
【論文タイトルと著者等】
タイトル:Environmental DNA metabarcoding on aquatic insects: Comparing the primer sets of MtInsects-16S based on the mtDNA 16S and general marker based on the mtDNA COI region
著者:Masaki Takenaka, Yuta Hasebe, Koki Yano, Seiya Okamoto, Koji Tojo, Masashi Seki, Shun Sekiguchi, Takeshi Jitsumasa, Nobuyuki Morohashi, Yoshihiro Handa, Tomoka Sakaba
掲載誌:Environmental DNA
掲載日:2024年7月18日(オンライン公開)
URL:https://doi.org/10.1002/edn3.588
D O I:10.1002/edn3.588
竹中將起(信州大学) 筆頭著者・責任著者
長谷部勇太(神奈川県環境科学センター)
谷野宏樹(自然科学研究機構・基礎生物学研究所)
岡本聖矢(土木研究所)
東城幸治(信州大学)
関将史(株式会社プラントビオ)
関口俊(株式会社プラントビオ)
實政武志(株式会社プラントビオ)
諸橋 伸行(株式会社生物技研)
半田 佳宏(株式会社生物技研)
坂場 友香(株式会社生物技研)
環境DNA解析は、水を汲み、濾過しただけの試料から、その環境に生息している対象とする生物相を把握することができる強力な生物モニタリング・ツールです。特定の環境における生物多様性の評価や、絶滅危惧種の生息の有無、その遺伝的多様性を評価する際にも有効です。水を汲むだけの非侵襲的サンプリングであり、生態系への影響も最小限です。
また、我々が開発したマーカーは、採水サンプルからの環境DNA解析(メタバーコーディング)による生物相の評価だけでなく、例えば、魚類の胃内容物や哺乳類の糞便試料から対象とする魚類や哺乳類の食性を網羅的に把握するようなメタバーコーディング解析にも応用可能であり、既に、こうした調査研究においても用いられ、強力なツールとして効果を発揮しています。本研究でも、陸生昆虫が多く検出されており、水生昆虫だけでなく、陸生昆虫を含めた昆虫類全般を対象とした網羅的解析も可能です。こうしたことから、近い将来におけるメタバーコーディング手法として汎用されることが期待されます。
【関連する論文】
本研究成果の筆頭著者・責任著者らによる先行研究(Takenaka M, Yano K, Suzuki T, Tojo K, 2023, Development of novel PCR primer sets for DNA barcoding of aquatic insects, and the discovery of some cryptic species, Limnology, 24: 121–136)において、昆虫類に汎用可能であるメタバーコーディング・マーカーを新規開発しており、本研究ではそのマーカーを実践的研究に用いることで、実用性の高さを実証した関係にある。Takenaka et al. (2023) は、論文公表以来、2000ダウンロードを超える多くのアクセスや引用(現時点で10論文に引用)があり、これらの反響の大きかさら、2024年10月に Limnology 誌の論文賞(Limnology Excellent Paper Award)を受賞しました。
【用語解説】
※1. 環境DNA 河川や湖沼などの環境中に浮遊する生物由来のDNAをさす。こうした環境中のDNAを解析することで、その環境中に生息している生物相を把握することができる。特定の種を対象に、対象種の生息の有無を評価する方法や、今回のように対象分類群(群集)を網羅的に解析する方法などが挙げられる。
※2. DNAバーコーディング 「標準化」された遺伝子領域(DNA断片)を対象に、データベースの登録配列を参照することで、迅速、正確、そして自動的に種を同定する概念。スーパーマーケットの製品コードである黒い縞模様(バーコード)をスキャナーで読み取る方法と似ていることから名付けられたもので、高い専門知識がなくても種同定が可能になるだけでなく、昆虫類の場合、幼虫や卵、不完全な標本(体の一部分など)からも種同定することができる。しかし、高い精度の分類学的根拠に基づくDNAデータベースの構築が必須。
※3. PCR ポリメラーゼ連鎖反応(Polymerase Chain Reaction)の略称で、対象とする遺伝子配列を増幅する手法。わずかなDNA量のサンプルであっても、特定の配列だけを様々な解析に使用できるように増幅させる。新型コロナウイルスの感染の有無を判定する際にも同じメカニズムを用いて、PCRによりウィルス特定の配列が増幅されるかどうかが検査された。
※4. PCRプライマー PCRにより特定のDNA配列を増幅させる際に、増幅させる遺伝子配列の両端に結合させて、DNAの増幅の開始点として機能させる20塩基程度の短い遺伝子断片。DNAに結合した2つのプライマーが、結合した箇所から互いにDNAの複製が進むことで、2つのプライマーの内側の配列が増幅される。新型コロナウイルスの感染の有無を検査するPCRにおいては、このウイルスに特異的なプライマーを設計することで、体内にウイルスが存在している時にのみその特定遺伝子配列が増幅される仕組みが利用された。
【論文タイトルと著者等】
タイトル:Environmental DNA metabarcoding on aquatic insects: Comparing the primer sets of MtInsects-16S based on the mtDNA 16S and general marker based on the mtDNA COI region
著者:Masaki Takenaka, Yuta Hasebe, Koki Yano, Seiya Okamoto, Koji Tojo, Masashi Seki, Shun Sekiguchi, Takeshi Jitsumasa, Nobuyuki Morohashi, Yoshihiro Handa, Tomoka Sakaba
掲載誌:Environmental DNA
掲載日:2024年7月18日(オンライン公開)
URL:https://doi.org/10.1002/edn3.588
D O I:10.1002/edn3.588
竹中將起(信州大学) 筆頭著者・責任著者
長谷部勇太(神奈川県環境科学センター)
谷野宏樹(自然科学研究機構・基礎生物学研究所)
岡本聖矢(土木研究所)
東城幸治(信州大学)
関将史(株式会社プラントビオ)
関口俊(株式会社プラントビオ)
實政武志(株式会社プラントビオ)
諸橋 伸行(株式会社生物技研)
半田 佳宏(株式会社生物技研)
坂場 友香(株式会社生物技研)
